生物医学研究是我们理解人类健康与疾病、药物发现和临床护理进展的基础。然而,随着复杂实验室实验、大型数据集、众多分析工具和海量文献的不断增长,生物医学研究日益受到重复性和碎片化工作流程的限制。Biomni是一种通用型生物医学人工智能代理,旨在自主执行跨多个生物医学子领域的广泛研究任务。为了系统地绘制生物医学动作空间,Biomni首先利用动作发现代理创建了首个统一的代理环境——从25个生物医学领域的数万篇出版物中挖掘关键工具、数据库和协议。在此基础上,Biomni采用了一种通用代理架构,该架构将(LLM)推理与检索增强规划和基于代码的执行相结合,使其能够动态组合和执行复杂的生物医学工作流程,完全无需依赖预定义模板或僵化的任务流程。系统基准测试表明,Biomni在异构生物医学任务(包括因果基因优先级排序、药物再利用、罕见病诊断、微生物组分析和分子克隆)中实现了强大的泛化能力,且无需任何特定任务的提示调优。
https://biomni.stanford.edu
背景概述
人工智能(AI)领域的最新进展已引发范式转变,为从根本上重塑生物医学研究创造了可能。通过自动化重复性任务、提高生产力,并实现此前难以想象的突破,AI Agent已显著重塑了软件工程、法律、材料科学和医疗保健等领域。鉴于这些发展,一个问题应运而生:我们能否构建一个虚拟的AI生物医学科学家?这样的虚拟科学家将自主处理跨越多个子领域的多样化生物医学研究任务,通过跨学科整合释放广泛能力并促成新见解——这一成就能够从根本上增强受限于专业知识的人类生物学家的能力。这种虚拟科学家能够高效管理数千个并发任务,可显著提升人类生产力并加快生物医学发现的步伐。
此前的方法在很大程度上依赖于为狭窄生物医学任务量身定制的专业代理工作流程(比如scGPT),这限制了它们在整个生物医学领域中灵活移动和进行泛化的能力,使AI Agent能够处理广泛的生物医学任务带来了巨大的技术挑战——最显著的是需要将高级推理能力与执行高度专业化生物医学操作的能力紧密结合。尽管基于大型语言模型(LLM)的推理已取得显著进展,但此类LLM需要访问一个明确界定生物医学动作空间的环境,而该空间本质上具有多样性、领域特异性和复杂性。此外,一个真正有能力的系统需要一种能够与该生物医学环境进行原生交互的代理架构——自主选择和组合动作,利用其推理能力规划和执行各种任务,而不依赖于僵化的预定义工作流程。
作为虚拟AI生物学家,Biomni可自主提出新颖且可验证的假设,执行复杂的生物信息学分析,并设计严谨的实验方案。为实现这一能力,首先通过系统分析从主要生物医学文献数据库中精选的、横跨25个不同子领域的数万篇生物医学研究论文,构建了一个统一且全面的生物医学动作空间。
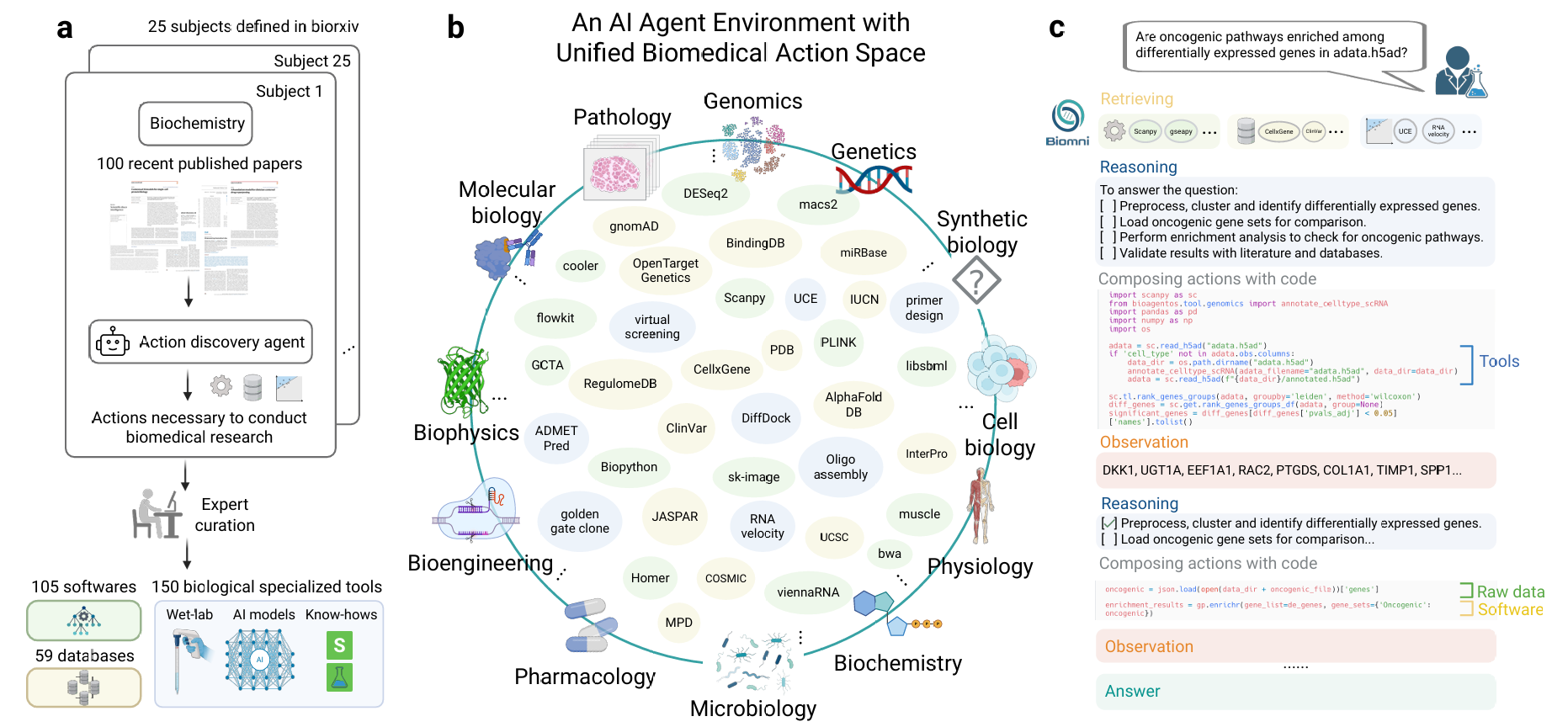
- 该动作空间主要由工具、数据库和软件等部分构成。其中,工具方面包含 150 种专业生物医学工具,这些工具为研究提供了具体的操作手段;数据库有 59 个,例如 PDB、OpenTarget、ClinVar 等,涵盖了蛋白质结构、基因靶点、遗传变异等多方面的数据,为研究提供了丰富的数据支持;软件有 105 个软件包 ,包括用于单细胞基因表达数据分析的 scanpy、进行基因集富集分析的 gseapy 等,满足了不同类型生物医学数据的分析需求。
在此基础上,作者开发了一个由大型语言模型(LLM)驱动的动作发现 agent,该agent能够阅读论文并提取推动生物医学发现所必需的关键任务、工具和数据库。这些元素随后被筛选并整合到Biomni-E1中,这是定义用于agent交互的生物医学动作空间的基础环境。Biomni-E1包含150种专业生物医学工具、105个软件包和59个数据库。随后,作者设计了Biomni-A1,这是一种通用agent架构,能够通过使用Biomni-E1提供的工具和数据集,灵活执行广泛的生物医学任务。当收到用户查询时,该agent首先使用检索系统识别所需的最相关工具、数据库和软件,然后应用基于LLM的推理和领域专业知识生成详细的分步计划。每个步骤都通过可执行代码表达,从而实现生物医学动作的精确且灵活的组合——考虑到该领域对高度专业化工具和数据资源的依赖,这是一项关键特性。与传统的函数调用方法不同,这种方法支持生物医学工作流程的动态性和复杂性。这种集成系统使Biomni不仅能够高效解决具有挑战性的大规模生物医学问题,还能泛化到生物医学研究中前所未见的新任务。
基准测试表明,Biomni在已建立的生物医学问答基准中表现出色,并且在开发过程中从未遇到的八个具有挑战性的现实场景中具有强大的泛化性能。此外,三个有影响力的案例研究突出了Biomni的实际能力:(1)分析458个可穿戴传感器文件数据以生成新见解;(2)对大规模原始数据集(如单细胞RNA-seq和ATAC-seq数据)快速进行全面的生物信息学分析,以生成新的见解和假设;(3)自主设计实验室协议以协助湿实验室研究人员。
Biomni概览
Biomni是一种通用型生物医学AI代理,由两个主要组件组成:Biomni-E1(一个具有统一动作空间的基础生物医学环境)和Biomni-A1(旨在有效利用该环境的智能agent)。
构建统一的生物医学动作空间具有挑战性,因其固有的复杂性和广泛性。作者通过采用AI驱动的方法系统性地解决这一问题(图1a)。具体而言,利用bioRxiv定义的25个学科类别,每个类别选取100篇最新的出版物。一个动作发现LLM代理依次处理每篇论文,提取复现或开展所述研究所需的关键任务、工具、数据库和软件。这组全面的资源构成了执行大量生物研究任务所需的基本动作。
- 图1:Biomni中的统一生物医学动作空间与agent概述。
- a) 系统构建统一生物医学动作空间的工作流程。使用AI驱动的发现 agent 从25个生物医学子领域的2500篇近期bioRxiv出版物中提取开展生物医学研究所需的动作。提取的动作经人类专家严格验证和筛选,最终整合了105个生物医学软件工具、150种专业生物工具(包括湿实验协议、AI驱动预测模型和领域特定知识)以及59个综合生物医学数据库。
- b) 统一生物医学动作空间的示意图,涵盖遗传学、基因组学、合成生物学、细胞生物学、生理学、微生物学、药理学、生物工程、生物物理学、分子生物学和病理学等多个生物医学子领域。图中展示了集成到Biomni环境中的代表性工具和数据库,突出其通用能力。
- c) 演示Biomni自主回答复杂生物学问题的推理与动作组合过程的示例工作流程。Biomni根据用户查询检索相关工具,制定结构化推理计划,并编写可执行代码以执行全面的生物信息学分析,基于观察结果迭代优化推理,直至得出最终精确答案。
随后作者精心构建了Biomni-E1,这是一个供生物医学AI agent执行广泛操作的环境(图1b)。所识别的工具均经过人类专家的严格验证,并配有相应的测试用例。这些工具(原论文补充表1至表18)因其非简易特性而被专门挑选,涵盖了复杂代码、领域特定知识或专业AI模型。考虑到生物软件所需的固有灵活性(无法始终简化为静态函数),作者构建了一个预安装105个常用生物软件包(原论文补充表23至表30)的执行环境,支持Python、R和Bash脚本。
在数据库方面,作者将资源分为两类。第一类包含可通过Web API访问的大型关系数据库(如PDB、OpenTarget、ClinVar)(原论文补充表19至表20)。作者没有创建大量单独的检索工具,而是为每个数据库实现了统一功能。每个功能接受自然语言查询,并在内部使用LLM解析数据库模式并动态生成可执行查询。没有Web接口的数据库被下载到数据池,并在本地预处理为结构化的pandas Dataframe,以便与agent无缝集成,Biomni-E1中共有59个数据库(原论文补充表21至表22)。总之,Biomni-E1是首个生物医学AI代理环境,包含150种专业生物医学工具、105个软件和59个数据库。
为了构建能够处理多样化生物医学任务的通用agent,我们需要一种专门的agent架构——避免为每个独立任务硬编码工作流程。这促使了Biomni-A1的开发,其整合了若干对跨生物医学研究领域操作至关重要的核心创新。首先,作者引入了基于大型语言模型(LLM)的工具选择机制,旨在应对生物医学工具的复杂性和专业性,可根据用户目标动态检索定制化的资源子集。其次,考虑到生物医学任务常需要丰富的程序逻辑,Biomni-A1将代码作为通用动作接口——使其能够组合和执行包含循环、并行化和条件逻辑的复杂工作流程。关键在于,这种方法还允许agent交错调用软件、工具、数据库和原始数据操作,而无需遵循预定义的函数签名,从而支持异构资源的灵活动态集成。第三,agent采用自适应规划策略:基于生物医学知识制定初始计划,并在执行过程中迭代优化,实现响应式、上下文感知的行为。这些创新共同使Biomni-A1能够泛化到前所未见的任务和领域,以体现通用生物医学智能的方式动态组合智能动作,并与软件、数据和工具交互(图1c)。
Biomni方法
从文献中发现动作
通过提取和解析PDF内容,收集并分析了bioRxiv上2024年的100篇近期出版物。每篇论文被分块处理,由一个专门的提示词引导大型语言模型(LLM)逐块处理,以明确识别和提取三类可操作的见解:任务,软件,数据库。具体而言,针对任务,LLM被指示突出显示生物医学研究工作流程中需要专门实现的重复性任务。
Biomni环境的实现
在环境构建的初始迭代中,作者采用了保守且有针对性的方法进行工具筛选。最初,任务是根据与主要研究兴趣(药物发现和临床生物医学)的相关性进行筛选的,保留的领域包括生物化学、生物工程、生物物理学、癌症生物学、细胞生物学、发育生物学、遗传学、基因组学、免疫学、微生物学、分子生物学、病理学、药理学、生理学、合成生物学和系统生物学。随后,这些任务被缩小到约1900个常见的重复性任务。这些任务进一步经过人工审查,以消除冗余并排除那些琐碎或可通过简单代码轻松实现的任务。我们强调选择需要大量领域专业知识的高度专业化任务,例如湿实验协议和高级AI模型。
随后,人类科学家与配备网络搜索能力的软件工程agent合作,实现了每个专业工具。每个工具都经过严格验证,要求成功通过明确界定的测试用例。这一严格流程最终形成了150个专业工具的精选集合。此外,还纳入了PubMed和Google Scholar等必要的文献检索工具,并为未来的迭代扩展做好了准备。
每个工具均通过全面的清单进行严格定义,该清单强制规定:(1)清晰且具描述性的名称;(2)详细的文档;(3)输出格式为详细的研究日志,并针对LLM解释进行优化;(4)包含并成功通过特定测试用例;(5)专业化标准——如果一项任务可通过LLM生成的简短代码轻松实现(例如简单的数据库查询),则不创建专用工具。
数据库被分类处理,对于可通过网络应用程序接口(API)访问的大型关系数据库(如蛋白质数据库PDB、OpenTargets、ClinVar等),作者通过统一的查询函数进行集成。该函数接受自然语言输入,并利用大型语言模型(LLM)动态解析数据库模式并执行相应查询。对于缺乏网络API的数据库,将其下载并在本地预处理为结构化的pandas数据,以便agent能够无缝访问。
在软件集成方面,考虑到经常需要同时使用多种软件工具,作者构建了一个统一的容器化环境,其中预安装了一整套全面的相关软件。此外,该环境还支持R软件包和命令行界面(CLI)工具的执行。
Biomni-A1
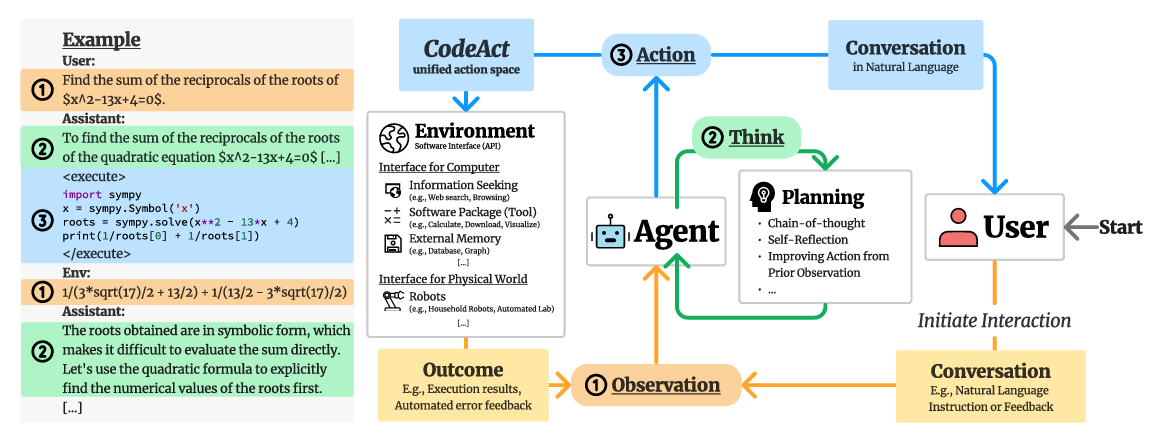
Biomni agent是一种基于CodeAct框架(Executable code actions elicit better llm agents)构建的通用生物医学AI代理,旨在通过将Agent(LLM)与环境(可交互式编码环境)相结合来系统解决生物医学任务。在收到用户查询时,Biomni首先促使LLM生成清晰的编号要点列表计划,详细列出解决给定问题所需的步骤,同时仔细跟踪进展和沿途的调整。由于工具、软件和数据库空间庞大,查询任务可能仅使用其中的一小部分资源。为避免上下文过长,系统利用一个独立LLM驱动的基于提示的检索器,使agent能够从可用资源中动态选择最相关的函数、数据集和软件库。在执行过程中,LLM生成代码并在编码环境(Python、R或Bash)中执行,然后返回结果观察以指导后续推理。这种迭代方法持续进行,直至代理得出准确且经过验证的解决方案。
Executable code actions elicit better llm agents,ICML2024
- 大型语言模型 (LLM) 代理能够执行各种操作,例如调用工具和控制机器人,在应对现实世界挑战方面展现出巨大潜力。LLM 代理通常通过生成 JSON 或预定义格式的文本来执行操作,但这通常受限于操作空间(例如,预定义工具的范围)和灵活性(例如,无法组合多个工具)。该研究提出使用可执行 Python 代码将 LLM agent的操作整合到一个统一的操作空间 (CodeAct)。CodeAct 与 Python 解释器集成,可以执行代码操作,并通过多轮交互动态修改先前的操作或根据新的观察结果发出新的操作。CodeAct对 API-Bank 和新整理的基准测试中的 17 个 LLM 进行了广泛的分析,结果表明 CodeAct 的性能优于广泛使用的替代方案(成功率最高高出 20%)。CodeAct 令人鼓舞的性能激励我们构建一个开源 LLM 代理,它通过执行可解释代码与环境交互,并使用自然语言与用户协作。为此,CodeAct 收集了一个指令调优数据集 CodeActInstruct,其中包含 7000 条多轮交互。我们证明,它可以与现有数据结合使用,在不影响模型通用能力的情况下,改进面向代理任务的模型。CodeActAgent 经过 Llama2 和 Mistral 的微调,与 Python 解释器集成,并经过专门定制,可以使用现有库执行复杂任务(例如模型训练),并能够自主调试。
- https://github.com/xingyaoww/code-act
- CodeAct:通用agent多轮交互框架,该框架描述了 CodeAct 的作用。CodeActInstruct 专注于agent与环境的交互,并专门筛选自我改进的规划行为,而通用对话数据则侧重于agent与用户的交互。(用户-agent-环境)